近日,必赢电子游戏网站田圃教授应邀在国际著名期刊WIREs Computational Molecular Science发表原创局部分布理论综述论文(The repetitive local sampling and the local distribution theory. WIREs Computational Molecular Science, 2021, e1588)。
深度学习在生物大分子静态结构预测中取得了巨大进展(Nature, 596:583-589, 2021; Science, 373:871-876, 2021; Science, 373:1047-1051, 2021),但这些架构都不能描述至关重要的构象变化。分子模拟是生物大分子体系全景动态行为描述的计算手段,但其“分子力场+采样”的传统框架最近二十多年一直陷于准确度和效率之间选择的困境。深度学习神经网络力场解决了传统分子力场固定函数形式和两两相互作用近似的局限,显著提高了模拟准确度,但也降低了效率。粗粒化方法提高了效率但降低了准确度。增强采样方法对反应坐标难以确定的复杂体系应用困难,这些研究方向的基础理论最初都在欧美提出。而达成典型分子生物体系的日常可靠智能设计预测需要在提高全原子模拟准确度的同时大幅度提高效率。因此亟需新理论和算法框架的突破。
在努力突破上述学科发展瓶颈的探索中,田圃团队发现了分子模拟中普遍存在的重复局部采样和伴随的计算资源浪费,并针对此问题提出了针对物质体系的全新统计机器学习理论-局部分布理论。简述如下,对一个有n个基本自由度的分子体系,其所有的行为由全局分布决定。为利用局部分布,我们可以把全局分布以局部变量集合的形式表达:
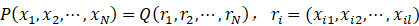 (1)
(1)
其中,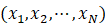 是基本自由度,
是基本自由度,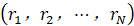 中的每个
中的每个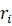 都是由以
都是由以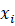 为中心的多个基本自由度组成的局部变量集合。继续做如下变换即得到局部分布理论:
为中心的多个基本自由度组成的局部变量集合。继续做如下变换即得到局部分布理论:
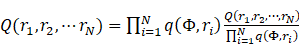
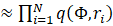 ,按照
,按照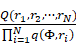 中的介导和全局关联约束采样 (2)
中的介导和全局关联约束采样 (2)
关键的步骤是把复杂的介导全局关联通过采样约束近似实现,如果有直接长程相互作用则加入相应的贡献。求解全局分布则是这个公式推导的逆过程,首先通过机器学习拟合局部分布,然后按照采样约束(和直接长程相互作用的修正)动态拼图得到全局分布。
针对蛋白质分子结构优化任务的核心是自由能最小化的本质,田圃团队首次引入计算图和自动微分,发展了局部分布理论的局部最大似然近似实现,并通过坐标变换实现了严格的键长键角刚性约束,得到了端到端动态模拟和自由能计算的人工智能方法(RSC Adv.2021,11:12929)。在CASP14 (https://www.predictioncenter.org/casp14/index.cgi)全球蛋白质结构优化竞赛中,如图1所示,在难度最高(GDT打分大于60)的两组目标中,在获得最高准确度积分的同时展示出比主流方法高出三个数量级以上的效率。打破了主流分子模拟框架在准确度和效率之间选择的困境。
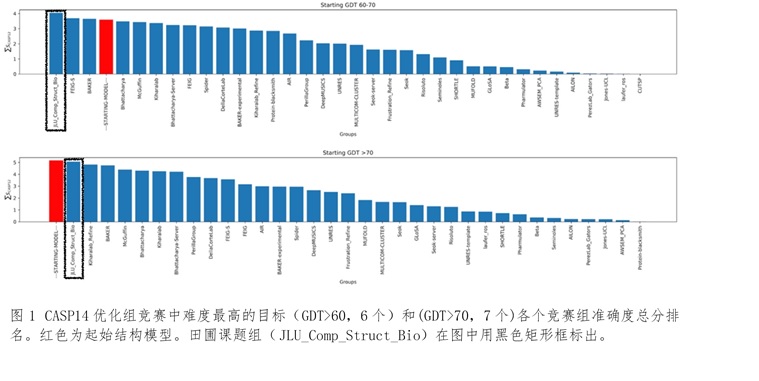
局部分布理论是一个广义统计机器学习理论,在蛋白质结构优化的初步验证中展示了其在全原子模拟基础上同时显著提高精度和效率的巨大潜力,原则上可以在准确完成相应局部分布的学习后适用于任意复杂分子体系。今后将进一步在多种生物以及化学和材料复杂分子体系发展该理论,以期在多个领域取代诸多昂贵费时的实验表征,革命性促进多学科复杂分子体系研究和工业化。
该论文唯一作者田圃教授,研究得到了国家重点研发计划(2017YFB0702500)的支持。
论文链接:https://wires.onlinelibrary.wiley.com/doi/full/10.1002/wcms.1588